انجماد الکترونی (cryo-ET) به عنوان یک تکنیک قدرتمند برای ارائه تصاویر سه بعدی دقیق از محیط های سلولی و زیست مولکول های محصور در حال ظهور است. با این حال، یکی از چالش های این روش، شناسایی مولکول های پروتئین در تصاویر برای پردازش بیشتر است. یک تیم تحقیقاتی پیرامون Stefan Raunser، مدیر MPI فیزیولوژی مولکولی در دورتموند، به رهبری تورستن واگنر، نرمافزاری را برای انتخاب پروتئینها در حجمهای شلوغ سلولی توسعه دادند. ابزار منبع باز جدید که TomoTwin نام دارد، مبتنی بر یادگیری متریک عمیق است و به دانشمندان اجازه می دهد تا چندین پروتئین را با دقت و توان عملیاتی بالا بدون ایجاد یا آموزش مجدد شبکه هر بار به صورت دستی پیدا کنند.
هرچی بیشتر بهتر
TomoTwin راه را برای شناسایی خودکار و محلی سازی پروتئین ها به طور مستقیم در محیط سلولی آنها هموار می کند و پتانسیل cryo-ET را گسترش می دهد.
گاوین رایس، نویسنده اول این نشریه
Cryo-ET این پتانسیل را دارد که نحوه کار مولکول های زیستی در سلول را رمزگشایی کند و از این طریق، اساس زندگی و منشاء بیماری ها را آشکار کند.
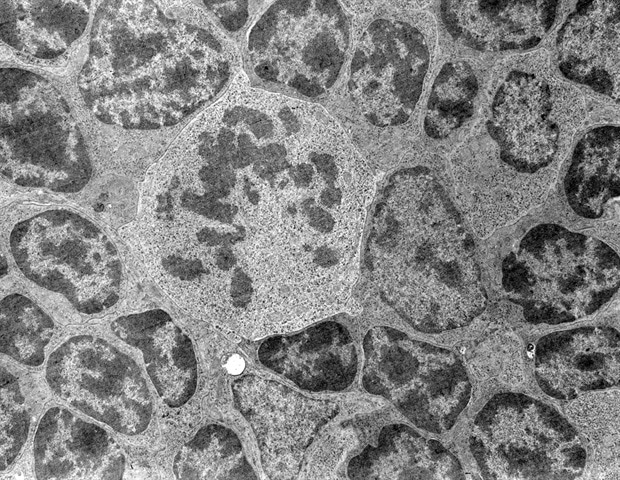
در یک آزمایش cryo-ET، دانشمندان از یک میکروسکوپ الکترونی عبوری برای به دست آوردن تصاویر سه بعدی، به نام توموگرام، از حجم سلولی حاوی مولکول های زیستی پیچیده استفاده می کنند. برای به دست آوردن تصویری با جزئیات بیشتر از هر پروتئین مختلف، آنها تا حد ممکن تعداد کپی از آنها را به طور میانگین می گیرند – مشابه عکاسانی که یک عکس را با نوردهی های مختلف می گیرند تا بعداً آنها را در یک تصویر کاملاً نوردهی شده ترکیب کنند. مهم این است که قبل از میانگین گیری پروتئین های مختلف در تصویر، باید به درستی شناسایی و مکان یابی شود. رایس میگوید: دانشمندان میتوانند روزانه صدها توموگرام به دست آورند، اما ما فاقد ابزارهایی برای شناسایی کامل مولکولهای درون آنها هستیم.
دستچین کردن
تاکنون، محققان از الگوریتمهایی مبتنی بر الگوهای ساختارهای مولکولی از قبل شناخته شده برای جستجوی منطبقها در توموگرامها استفاده میکردند، اما این الگوریتمها مستعد خطا هستند. شناسایی مولکول ها با دست گزینه دیگری است که انتخاب با کیفیت بالا را تضمین می کند اما برای هر مجموعه داده از روزها تا هفته ها طول می کشد.
امکان دیگر استفاده از نوعی یادگیری ماشینی تحت نظارت است. این ابزارها میتوانند بسیار دقیق باشند، اما در حال حاضر فاقد قابلیت استفاده هستند، زیرا برای آموزش نرمافزار برای هر پروتئین جدید، نیاز به برچسبگذاری دستی هزاران نمونه دارند، کاری تقریبا غیرممکن برای مولکولهای بیولوژیکی کوچک در یک محیط سلولی شلوغ.
TomoTwin
نرم افزار تازه توسعه یافته TomoTwin بر بسیاری از این موانع غلبه می کند: می آموزد که مولکول هایی را که از نظر شکل مشابه در یک توموگرام هستند انتخاب کند و آنها را به یک فضای هندسی ترسیم کند – سیستم برای قرار دادن پروتئین های مشابه در نزدیکی یکدیگر پاداش می گیرد و در غیر این صورت جریمه می شود. در نقشه جدید، محققان میتوانند پروتئینهای مختلف را جداسازی و به دقت شناسایی کنند و از آن برای قرار دادن آنها در داخل سلول استفاده کنند. رایس می گوید: «یکی از مزیت های TomoTwin این است که ما یک مدل چیدن از پیش آموزش دیده ارائه می دهیم. با حذف مرحله آموزش، نرم افزار حتی می تواند بر روی رایانه های محلی اجرا شود – جایی که پردازش توموگرام معمولاً به 60 تا 90 دقیقه نیاز دارد، زمان اجرا در ابررایانه MPI Raven به 15 دقیقه در هر توموگرام کاهش می یابد.
TomoTwin به محققان این امکان را می دهد که ده ها توموگرام را در مدت زمانی که برای انتخاب دستی یک توموگرام لازم است انتخاب کنند، بنابراین برای به دست آوردن تصویر بهتر، توان عملیاتی داده ها و نرخ میانگین را افزایش می دهند. این نرم افزار در حال حاضر می تواند پروتئین های کروی یا کمپلکس های پروتئینی بزرگتر از 150 کیلودالتون را در سلول ها پیدا کند. در آینده، هدف گروه Raunser شامل پروتئین های غشایی، پروتئین های رشته ای و پروتئین هایی با اندازه های کوچکتر است.
منبع:
موسسه فیزیولوژی مولکولی ماکس پلانک
مرجع مجله:
برنج، جی. و همکاران. (2023). TomoTwin: محلی سازی 3 بعدی تعمیم یافته ماکرومولکول ها در توموگرام های کریو الکترونی با داده کاوی ساختاری. روش های طبیعت. doi.org/10.1038/s41592-023-01878-z.
